【受付時間】 10:00〜18:00(土日祝日を除く)
【受付時間】 10:00〜18:00
(土日祝日を除く)

GATTAquant
超解像イメージングのための革新的なツール 「ナノルーラー」とは?
| 製品カテゴリー | 超解像顕微鏡 解像度評価ツール ナノルーラー |
|---|
回折限界 超えた解像度の評価:ナノルーラー
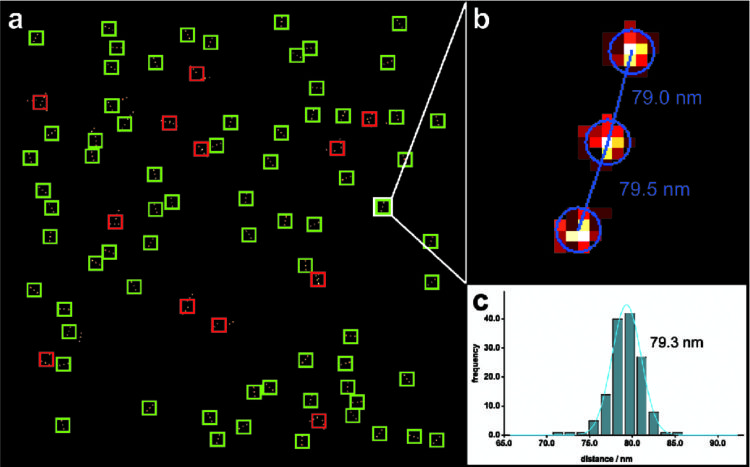
回折限界 を打破した超解像技術がいくつも発明されたことで、蛍光顕微鏡法における空間的分解能は目覚ましい進歩を遂げました。しかしながら、信頼のおけるツールがなかったことで、これらの新規技術の解像度が正しいものなのかを確認するのは困難でした。新たに開発されたナノ構造「ナノルーラー」は、2つの蛍光マーク間の距離が正確に定められ、色素分子をナノメートルレベルの正確さで配置することができ、ほぼ均一に製造することができます。
ナノルーラーが誕生した背景
21世紀最初の10年間は、光学顕微鏡の目覚ましい向上が見られた時期でした。光学的な回折限界は1873年にエルンスト・アッベにより定義されましたが、最高6ナノメートルの空間分解能を有する超解像技術が複数発明されたことでこの限界が打破されました。向上した空間分解能を達成するために超解像顕微鏡のユーザーが追う負担、つまりデータ解析、セットアップの複雑さや試料の準備といった作業の複雑さは日増しに高まっています。このため、顕微鏡のハード的なスペックだけではこれら新技術の空間分解能を計算することは、もはや不可能となっています。
空間分解能は、原則的にあらゆる顕微鏡の最も重要な性能ですので、ユーザー、メーカー、開発者にとって空間分解能を求めることは極めて重要です。従来は、微小管やアクチンフィラメントといった細胞構造をイメージングすることで空間分解能の検証および測定が行われていました。しかしこれらの試料では、確認したい大きさのサンプルがなかったり、標識濃度や色素間の距離均一性が低いといったデメリットがあります。これらの問題点により、空間解像度を検査するための信頼できる手法はありませんでした。
DNAナノテクノロジーに基づいたナノルーラー
上述の全ての要求を満たす検査試料は、「DNA折り紙」技術により作成された「ナノルーラー」です。DNAオリガミを端的に述べますと、ナノスケールのブレッドボードとして考えることができ、色素分子、蛍光タンパクあるいは一本鎖DNAに結合できあらゆる物質を配置することができます。DNA折り紙構造は高度に均一に製造することができ、自己集合します。そのため、ユビキタスに利用可能な試料として使用することができます。DNA折り紙に基づいた、ナノルーラーは、図1でご覧いただけます。 通常、いくつかの有機色素分子から成る蛍光マークが各DNA折り紙構造上に2個から3個ずつ配置されます。これら蛍光マーク間の距離は、6~360nmの範囲で自由に調整することができます。蛍光マークごとの色素分子の数は10~20の範囲にあり、一般に使用されている蛍光色素であれば基本的にナノルーラーに取り付けることができます。ナノルーラーはすぐに使える状態で製造することができ、DNA折り紙構造をμm2あたり約1個の濃度でカバースリップ上に固定します。つまり通常用いられる視野において数百個のナノルーラーが観測されます。これにより蛍光マーク間の距離を測定することができ、顕微鏡の空間分解能を検査し、測定の統計的エラーを計算できるようになります。同一の構造が大量にあるため、ナノルーラーを自動的に検出する効率的なアルゴリズムを使用でき、蛍光マーク間の距離を測定できるようになります(図2)。すぐに使える状態のナノルーラー試料は冷蔵庫内で数か月間保存することができ、日常的に空間分解能を測定できるようになります。図1にあるように、ナノルーラーは従来の共焦点顕微鏡だけでなく、STED、SIMや、あるいはdSTORM、PALM、GSDIM、DNA-PAINTなどの局在化法などすべての超解像技術に最適化されています。研究室においてナノルーラーは、イメージングを最適化したり、超解像の原理を講義したり、顕微鏡が問題なく稼働しているかを判定したり、あるいはどの設定が研究の要求に最も適しているかを決定したりするのに使用されます。顕微鏡メーカーは、新規技術や新製品の開発だけでなく、自社製品が技術的な仕様や要求される空間分解能を満たしていることを実証するためにナノルーラーを用いています。
結論
超解像蛍光顕微鏡の新手法により、空間分解能が10倍ほど向上しています。しかし、生体試料の準備、顕微鏡の操作、データ解析には大変な労力がかかり、正確な空間分解能を計算することはもはや不可能となっています。DNA折り紙に基づいたナノルーラーは、蛍光マーク間の距離が一定であり、標準的なカバースリップ上で大量に固定できるという、理想的な検査試料です。ナノルーラーにより、従来の共焦点顕微鏡だけでなく、現代の超解像顕微鏡の空間的分解能を測定することができます。
FAQ
-
カタログにある蛍光色素を変えることはできるか?
可能です。励起波長及び差し支えない範囲で顕微鏡の情報を教えてください。
-
蛍光マークの距離を変えることはできるか?
可能です。蛍光マーク間の距離が6~350nmの間で変更できます。ご希望の条件に合わせ、ベストな距離をお選びいただけます。
-
GATTAquant製品は、発注から納品までどのくらいかかるか?
標準品の場合、通常は約3~4週間かかります。
特注の場合、約5~7週間です。 -
製品はどのように納品されるか?
冷蔵便あるいは製品タイプによっては冷凍便で配送します。
-
GATTAquant製品はどんな見た目か? またどの種類のカバースリップが使われているか?
フローチャンバーは3×1インチ、厚さ1mmの商用対物スライドの上にマウントされます。対物スライドにはGATTAquantのロゴと、サンプルの情報を示すスティッカーラベルがスライドの反対側に描かれています。カバースリップのサイズは22×22mm2ですが、ご希望により18×18mm2のカバースリップもご提供できます。当社は厚さ170±5μmのMarienfeld High Precision coverslips #1.5のみを使用しています。スライドを顕微鏡に乗せる際、カバースリップが対物レンズの方を向いているかご確認下さい(たとえば、倒立顕微鏡を使う場合、カバースリップは対物レンズに向かって下向きになります。その際、ロゴとラベルは読める向きになります)。特別なカバースリップや異なるサイズ、あるいは他のカスタムをご希望の場合は、ご連絡ください。)
-
DNA-PAINTとは何か?
DNA-PAINTとは局在化技術に基づいた超解像顕微鏡です。ブリンキング(明減)を生み出すために、蛍光標識したオリゴヌクレオチドのDNA折り紙構造への一過性の結合を利用します。(Jungmann, R.; Steinhauer, C.; Scheible, M. B.; Kuzyk, A.; Tinnefeld, P.; Simmel, F. C. Single-Molecule Kinetics and Super-Resolution Microscopy by Fluorescence Imaging of Transient Binding on DNA Origami.Nano Lett 2010, 10, 4756–4761)。蛍光標識したオリゴヌクレオチドはイメージングバッファー内で拡散しますので、試料のイメージングはTIRFモードで行ってください。エピモードでDNA-PAINT試料をイメージングすると非常に強いバックグラウンドノイズを生じ、SN比が非常に小さくなります。通常、当社のGATTA-PAINTあるいはGATTA-PAINT HiResナノルーラーを局在化顕微鏡とともに使用することを推奨いたします。dSTORMを使い試験したい場合は、STORMナノルーラーを推奨します。
-
dSTORMナノルーラーとDNA-PAINTナノルーラーの違いはか?
STORMナノルーラーは、dSTORM用に作成されています。dSTORM技術は有機色素分子、たとえばグルコースオキシダーゼ、カタラーゼ、βメルカプトエタノールを含むバッファー中のAlexa Flour 647を使用します。こちらもご参照ください。Rust, M. J.; Bates, M.; Zhuang, X. Sub-Diffraction-Limit Imaging by Stochastic Optical Reconstruction Microscopy (STORM). Nat Meth 2006, 3, 793–795.STORMナノルーラーとDNA-PAINTナノルーラーは、ともに局在化法顕微鏡の空間分解能の検査を目的としています。二つの違いは、本質的には色素分子のオン-オフの切り替えを生み出す方法です。通常、DNA-PAINTあるいはDNA-PAINT HiResナノルーラーを推奨いたします。このタイプは数か月間保存可能であり、製造後すぐに使用可能です。試料の保存性の高さに加え、DNA-PAINTは光安定性、光子カウントが向上しており、従って解像度の向上が期待できます。dSTORM機構を使用してお使いの顕微鏡の検査をしたい場合は、STORMナノルーラーを推奨いたします。
-
DNA-PAINT HiResナノルーラーの画像には、どのように横変位の補正を行えばよいか?
DNA-PAINT HiResナノルーラーは、横変位の補正用の基準点として使用する基準マーカー(fiducial marker)を含んでいます。ソフトウェアであるGATTAnalysisを使用して補正ができます。
-
ソフトウェア: GATTAnalysis ライセンスファイルがソフトウェアに認識されない。どのような理由が考えられるか?
ソフトウェアをzipファイル外や、ネットワークリソースから起動させた場合はライセンスファイルが正しく認識されないことがあります。この場合、ソフトウェアおよびライセンス両方のファイルを抽出し、お使いのコンピューター上のローカルファイルに保存してから、もう一度ソフトウェアを起動させてください。
-
GATTAnalysisではどのようにマルチカラーSTEDやSIMナノルーラーを解析できるか?
マルチカラー試料では、GATTAnalysisは両方のカラーチャンネルの距離を個別に計算できます。チュートリアルに詳細な解説があります。
参考文献
- 27.
- 26.
Marsh RJ., Pfisterer K., Bennett P., Hirvonen LM., Gautel M., Jones GE., Cox S. (2018): Artifact-free high-density localization microscopy analysis – Nature Methods https://doi.org/10.1038/s41592-018-0072-5
- 25.
Mailfert S., Touvier J., Benyoussef L., … Bertaux N. (2018): A Theoretical High-Density Nanoscopy Study Leads to the Design of UNLOC, a Parameter-free Algorithm – Biophysical Journal https://doi.org/10.1016/j.bpj.2018.06.024
- 24.
Raab, M., Jusuk, I., Molle, J., Buhr, E., Bodermann, B., Bergmann, D., Bosse, H., Tinnefeld, P. (2018): Using DNA origami nanorulers as traceable distance standards and nanosocopic benchmark structures – Sci Rep, 8, 1
- 23.
Tortarolo, G., Castello, M., Diaspro, A., Koho, S. & Vicidomini, G. (2018): Evaluating image resolution in stimulated emission depletion microscopy – Optica, 5, 1
顕微鏡タイプ:STED - 22.
Göttfert, F. et al. (2017): Strong signal increase in STED fluorescence microscopy by imaging regions of subdiffraction extent – Proc Natl Acad Sci USA doi:10.1073/pnas.1621495114
顕微鏡タイプ:STED - 21.
K. Korobchevskaya, H. Colin-York, C. Lagerholm, M. Fritzsche (2017): Exploring the Potential of Airyscan Microscopy for Live Cell Imaging – Photonics, 4, 41
- 20.
Q. Wei, G. Acuna, S. Kim, C. Vietz, D. Tseng, J. Chae, D. Shir, W. Luo, P. Tinnefeld, A. Ozcan (2017): Plasmonics Enhanced Smartphone Fluorescence Microscopy – Sci Rep, 7, 2124
- 19.
R. Lin, A. Clowsley, I. Jayasinghe, D. Baddeley, C. Soeller (2017): Algorithmic corrections for localization microscopy with sCMOS cameras – characterisation of a computationally efficient localization approach – Opt Express, 25, 11701-11716
- 18.
S. Tajada, C. Moreno, S. O’Dwyer, S. Woods, D. Sato, M. Navedo, L. Santana (2017): Distance constraints on activation of TRPV4 channels by AKAP150-bound PKCa in arterial myocytes – J Gen Physiol, 149, 639-659
- 17.
M. Schropp, C. Seebacher, R. Uhl (2017): XL-SIM: Extending Superresolution into Deep Layers – Photonics, 4, 33
- 16.
R. Diekmann, Ø. Helle, C. Øie, P. McCourt, T. Huser, M. Schüttelpelz, B. Ahluwalia (2017): Chip-based wide field-of-view nanoscopy – Nature Photonics
- 15.
R. Lin, A. H. Clowsley, I. D. Jayasinghe, D. Baddeley, C. Soeller (2017): Algorithmic corrections for localization microscopy with sCMOS cameras – characterisation of a computationally efficient localization approach – Opt. Express, 25, 11701-11716
顕微鏡タイプ:PALM - 14.
I. M. Antolovic, S. Burri, C. Bruschini, R. A. Hoebe, E. Charbon (2017): SPAD imagers for super resolution localization microscopy enable analysis of fast fluorophore blinking – Scientific Reports, 7, 44108
顕微鏡タイプ:PALM - 13.
M. Heilemann, F. Fricke, C. Karathanasis, G. Hummer (2017): Molecule counts in localization microscopy with organic fluorophores – ChemPhysChem
顕微鏡タイプ:PALM - 12.
M. Dienerowitz, T. Heitkamp, T. Gottschall, J. Limpert, M. Borsch (2017): Confining Brownian motion of single nanoparticles in an ABELtrap – arXiv.org
- 11.
F. Göttfert, T. Pleiner, J. Heine, V. Westphal, D. Görlich, S. J. Sahl, S. W. Hell (2017): Strong signal increase in STED fluorescence microscopy by imaging regions of subdiffraction extent – PNAS
顕微鏡タイプ:STED - 10.
F. Balzarotti, Y. Eilers, K. C. Gwosch, A. H. Gynna, V. Westphal, F. D. Stefani, J. Elf, S. W. Hell (2016): Nanometer resolution imaging and tracking of fluorescent molecules with minimal photon fluxes – Science
- 9.
T. J. Lambert, J. C. Waters (2016): Navigating challenges in the application of superresolution microscopy – J Cell Biol
- 8.
S. C. Sidenstein, E. D’Este, M. J. Böhm, J. G. Danzl, V. N. Belov, S. W. Hell (2016): Multicolour Multilevel STED nanoscopy of Actin/Spectrin Organization at Synapses – Scientific Reports, 6, 26725
顕微鏡タイプ:STED - 7.
B. J. Glasgow (2016): Conventional fluorescence microscopy below the diffraction limit with simultaneous capture of two fluorophores in DNA origami – Proc. SPIE9714
- 6.
S. Schedin-Weiss, I. Caesar, B. Winblad, H. Blom, L. O. Tjernberg (2016): Super-resolution microscopy reveals γ-secretase at both sides of the neuronal synapse – Acta Neuropathologica Communications, 4:29
- 5.
R. T. Borlinghaus, C. Kappel (2016): HyVolution—the smart path to confocal super- resolution – Nature Methods, 13
顕微鏡タイプ:共焦点顕微鏡 - 4.
I. Gyongy, A. Davies, N. Dutton, R. Duncan (2016): Smart-Aggregation Imaging for Single Molecule Localization with SPAD Cameras – arXiv.org
- 3.
N. Chiaruttini, L. Redondo-Morata, A. Colom, F. Humbert, M. Lenz, S. Scheuring, A. Roux (2015): Relaxation of Loaded ESCRT-III Spiral Springs Drives Membrane Deformation – Cell, 163, 1-14
- 2.
J. Huff, W. Bathe, R. Netz, T. Anhut, K. Weisshart, Carl Zeiss Microscopy GmbH (2015): Confocal Imaging with improved Signal-to-Noise Ratio and Superresolution – The Airyscan Detector from ZEISS
- 1.
C. Kappel, I. Köster, Leica Microsystems (2015): Increasing Confocal Resolution Down to 140 nm – HyVolution Confocal Super-Resolution Reveals More Details in Crisp Images – Science Lab
GATTAquant社及びパートナーの共同論文及びアプリケーションノート
- 14.
Scheible, M.B., Tinnefeld, P. (2018): Quantifying Expansion Microscopy with DNA Origami Expansion Nanorulers – bioRxiv
顕微鏡タイプ:Expansion Microscopy - 13.
B. Eggart, M. Scheible, C. Forthmann (2017): Beyond the Diffraction Limit – Optik & Photonik, 2, 26
- 12.
J.J. Schmied, R. Dijkstra, M. Scheible, G. M. R. De Luca, J. J. Sieber, GATTAquant GmbH, Scientific Volume Imaging (SVI), Leica Microsystems (2016): Measuring the 3D STED PSF with a new Type of Fluorescent Beads – Science Lab
顕微鏡タイプ:STED - 11.
B. Eggart, M. Scheible, C. Forthmann (2016): Using Super-Resolution Nanorulers to study the Capabilities of EM-CCD and sCMOS Cameras beyond the Diffraction Limit – Hamamatsu Aplication Note
- 10.
J.J. Schmied (2016): Testing and Pushing the Limits of Super-Resolution Microscopy – Optik & Photonik, 11, 23-26
- 9.
Ta, H., J. Keller, M. Haltmeier, S. K. Saka, J. Schmied, F. Opazo, P. Tinnefeld, A. Munk, S. W. Hell (2015): Mapping molecules in scanning far-field fluorescence nanoscopy – Nature Commun., 6, 7977
顕微鏡タイプ:STED - 8.
J.J. Schmied, C. Forthmann, M. Scheible, GATTAquant GmbH (2015): Innovative Tools for Fluorescence Microscopy – Imaging & Microscopy, 17, 20-21
- 7.
J.J. Schmied, C. Forthmann, T. Straube, GATTAquant GmbH, Leica Microsystems (2015): Quantifying the Resolution of a Leica SR GSD 3D Localization Microscopy System with 2D and 3D Nanorulers – Science Lab
顕微鏡タイプ:STED - 6.
J.J. Schmied, M. Raab, C. Forthmann, E. Pibiri, B. Wünsch, T. Dammeyer, P. Tinnefeld (2014): DNA origami based standards for quantitative fluorescence microscopy – Nature Prot., 9, 1367–1391.
- 5.
A. Kurz, J.J. Schmied, K. Grußmayer, P. Holzmeister, P. Tinnefeld, D.-P. Herten (2013): Counting Fluorescent Dye Molecules on DNA Origami by Means of Photon Statistics – Small, 9 (23), 4061-8.
- 4.
J.J. Schmied, C. Forthmann, E. Pibiri, B. Lalkens, P. Nickels, T. Liedl, P. Tinnefeld (2013): DNA Origami Nanopillars as Standards for Three-dimensional Superresolution Microscopy – Nano Letters, 13 (2), 781–785.
- 3.
J.J. Schmied, A. Gietl, Phil Holzmeister, C. Forthmann, C. Steinhauer, T. Dammeyer, P. Tinnefeld (2012): Fluorescence and Super-resolution Standards based on DNA Origami – Nature Methods, 9, 1133–1134.
- 2.
Ralf Jungmann, Christian Steinhauer, Max Scheible, Anton Kuzyk, Philip Tinnefeld and Friedrich C. Simmel (2010): Single-Molecule Kinetics and Super-Resolution Microscopy by Fluorescence Imaging of Transient Binding on DNA Origami. – Nano Letters, 11:2475-2490.
- 1.
Christian Steinhauer, Ralf Jungmann, Thomas L. Sobey, Friedrich C. Simmel and Philip Tinnefeld (2009): DNA origami as a nanoscopic ruler for super-resolution microscopy. – Angew Chem Int Ed, 48(47):8797 – 8999.
超解像顕微鏡 解像度評価ツール ナノルーラー 関連商品のご紹介
お客様用ご相談/お問い合わせ
製品に関するご相談、デモ機器、お見積・納期などに関して お気軽に問い合わせ下さい
※は入力必須項目となります。
INFORMATIONお知らせ
-
2024.05.20
お知らせ
-
2024.02.01
新製品
-
2023.12.01
新製品
-
2023.08.21
新製品
-
2023.05.15
新製品